Wykorzystując sztuczną inteligencję naukowcy z Massachusetts Institute of Technology odkryli całkiem nową klasę związków, które mają ogromny potencjał w walce z infekcjami wywołanymi lekoopornymi bakteriami. Substancje te, jak wykazały testy, radzą sobie m.in. z MRSA i wykazują bardzo niską toksyczność względem ludzkich komórek.
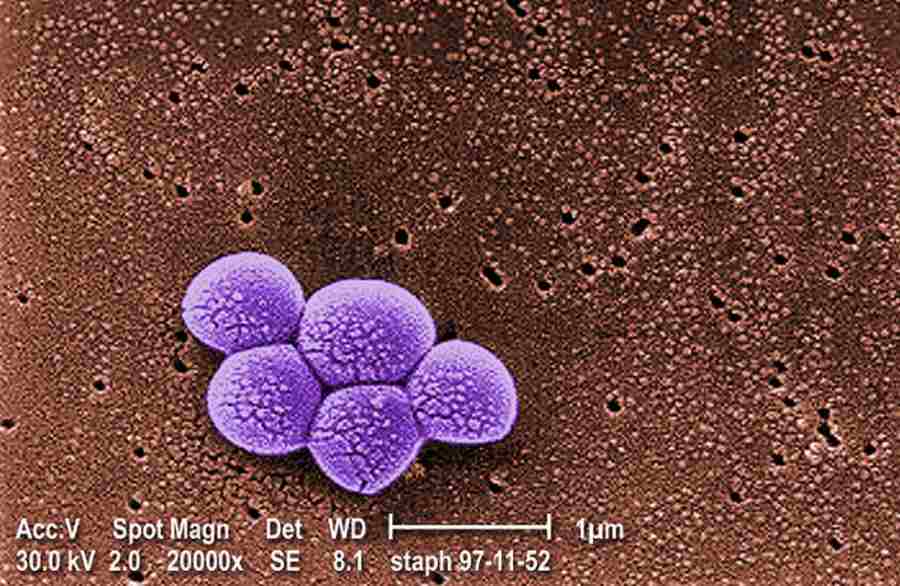
W badaniu opublikowanym na łamach pisma „Nature” (DOI: 10.1038/s41586-023-06887-8) naukowcy opisali nową klasę antybiotyków odkrytą przy pomocy algorytmów sztucznej inteligencji. Uczeni wykazali, że związki te radzą sobie z hodowanym w laboratorium gronkowcem złocistym opornym na metycylinę, czyli MRSA. Substancje te okazały się również skuteczne w dwóch mysich modelach zakażenia MRSA oraz w przypadku enterokoków opornych na wankomycynę (VRE). Co więcej, związki te wykazują również bardzo niską toksyczność wobec komórek ludzkich, co czyni je szczególnie dobrymi kandydatami na leki.
Związki opisane w publikacji pomogła zidentyfikować sztuczna inteligencja. Istotne w tym badaniu jest to, że uczeni byli także w stanie dowiedzieć się, jakiego rodzaju informacje wykorzystał model uczenia maszynowego do oszacowania siły działania nowych antybiotyków. Wiedza ta może pomóc naukowcom w opracowaniu dodatkowych leków, które mogą działać jeszcze lepiej niż te zidentyfikowane w algorytmy.
- Mogliśmy zobaczyć, czego nauczyły się modele, aby przewidzieć, że określone cząsteczki będą dobrymi antybiotykami. Nasza praca zapewnia ramy, które oszczędzają czas i zasoby – mówi James Collins z MIT.
Collins wraz ze swoim zespołem od kilku lat prowadzi program, w którym próbuje wykorzystać sztuczną inteligencję do wykrycia czy opracowania skutecznych środków przeciwko niosącym coraz większe zagrożenie lekoopornym patogenom. Ich prace przyniosły pierwsze rezultaty początkiem 2020 roku. Wówczas ukazała się praca, w której Collins ze współpracownikami opisali nowy, silny antybiotyk, który wykazuje dużą skuteczność przeciwko Acinetobacter baumannii oraz innym groźnym bakteriom (więcej na ten temat w tekście: Sztuczna inteligencja odkrywa nowe antybiotyki).
Związki te zidentyfikowano za pomocą modeli uczenia maszynowego. Mogą one nauczyć się identyfikować struktury chemiczne powiązane z działaniem przeciwdrobnoustrojowym. Modele te przesiewają miliony związków, generując przewidywania, które z nich mogą mieć silne działanie przeciwbakteryjne.
Tego typu wyszukiwania okazały się owocne, ale jednym z ograniczeń takiego podejścia jest to, że modele nie zdradzają, na podstawie jakich informacji oparły swoje przewidywania. -Modele te składają się z bardzo dużej liczby obliczeń imitujących połączenia neuronowe i nikt tak naprawdę nie wie, co się dzieje pod maską – powiedział Felix Wong, współautor publikacji.
Najpierw badacze przeszkolili model głębokiego uczenia, korzystając ze znacznie rozszerzonych zbiorów danych. Dane szkoleniowe wygenerowali na podstawie testów około 39 tys. związków, które sprawdzano pod kątem aktywności przeciwko MRSA. Następnie wprowadzili te dane oraz informacje o strukturze chemicznej związków do modelu sztucznej inteligencji.
- W zasadzie można przedstawić dowolną cząsteczkę jako strukturę chemiczną, a także określić model, czy ta struktura chemiczna jest antybakteryjna, czy nie. Model jest szkolony na wielu takich przykładach. Jeśli następnie dasz mu jakąkolwiek nową cząsteczkę, nowy układ atomów i wiązań, możesz określić prawdopodobieństwo, czy ten związek ma działanie antybakteryjne – przyznał Wong.
Aby dowiedzieć się, w jaki sposób model dokonuje swoich przewidywań, badacze zaadaptowali inny algorytm, który został wcześniej wykorzystany do wyjaśnienia innych modeli głębokiego uczenia, takich jak AlphaGo. Ten algorytm pozwala modelowi nie tylko wygenerować oszacowanie aktywności przeciwdrobnoustrojowej każdej cząsteczki, ale także przewidzieć, które podstruktury cząsteczki prawdopodobnie odpowiadają za tę aktywność.
Ale to nie wszystko. Naukowcy przeszkolili trzy dodatkowe modele głębokiego uczenia, aby przewidzieć, czy związki będą toksyczne dla trzech różnych typów ludzkich komórek. Łącząc te informacje z przewidywaniami dotyczącymi aktywności przeciwdrobnoustrojowej, wytypowali związki, które mogą zabijać drobnoustroje, wywierając jednocześnie minimalny niekorzystny wpływ na organizm.
Naukowcy uzbrojeni w kilka modeli sztucznej inteligencji sprawdzili około 12 milionów związków. Wśród nich modele zidentyfikowały substancje z pięciu różnych klas, w oparciu o podstruktury chemiczne w cząsteczkach, które według przewidywań były aktywne przeciwko MRSA.
Naukowcy przetestowali 280 związków pod kątem MRSA hodowanego w naczyniu laboratoryjnym, co pozwoliło im zidentyfikować dwa, które okazały się bardzo obiecującymi kandydatami na antybiotyki. W testach na dwóch modelach mysich, jednym dotyczącym zakażenia skóry MRSA i drugim zakażenia ogólnoustrojowego MRSA, każdy z tych związków zmniejszał populację MRSA 10-krotnie.
- Mamy dość mocne dowody na to, że ta nowa klasa strukturalna jest aktywna przeciwko patogenom Gram-dodatnim. Cząsteczki atakują selektywnie błony komórkowe bakterii w sposób, który nie powoduje znacznych uszkodzeń błon komórkowych człowieka. Nasze znacznie ulepszone podejście oparte na głębokim uczeniu pozwoliło przewidzieć tę nową klasę strukturalną antybiotyków i umożliwiło stwierdzenie, że nie jest ona toksyczna w stosunku do komórek ludzkich – podkreślił Wong.
Laboratorium Collinsa pracuje już nad zaprojektowaniem dodatkowych kandydatów na leki w oparciu o wyniki nowego badania, a także wykorzystaniem modeli do poszukiwania związków, które mogą zabijać inne typy bakterii.
Źródło: Massachusetts Institute of Technology, fot. CDC/ CC0